Unterschied zwischen QTL und GWAS
Der Schlüsselunterschied Zwischen QTL und GWAS beruht auf der Art der in der Analyse verwendeten Sequenzen. QTL verwendet Verknüpfungsgenorte, um phänotypische Merkmale zu analysieren, die mit polygener Vererbung assoziiert sind, während GWAS Ganzgenomsequenzen verwendet, um einzelne Nukleotidpolymorphismen einer bestimmten Erkrankung zu analysieren.
QTL -Karten und GWAS spielen eine wichtige Rolle bei den genetischen Analysen für verschiedene Merkmale. Darüber hinaus sind sie wichtig, um verschiedene Erkrankungen mit unbekannter Ätiologie zu analysieren. Darüber hinaus spielt die Sequenzierung eine Schlüsselrolle bei beiden Techniken. Diese Techniken können weiter mit anderen Hochdurchsatztechniken gekoppelt werden, um eine erhöhte Genauigkeit und Präzision zu erhöhen.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist Qtl
3. Was ist GWAS
4. Ähnlichkeiten zwischen QTL und GWAS
5. Seite an Seite Vergleich - Qtl vs Gwas in tabellarischer Form
6. Zusammenfassung
Was ist Qtl?
Qtl steht für Quantitativer Merkmal Locus. Es ist eine Region der DNA, die mit einem phänotypischen Merkmal assoziiert ist. Im Allgemeinen führt ein QTL zu polygenen Effekten. Die Verteilung von QTLs variiert, und die Anzahl der QTLs legt den Grad der Variation eines bestimmten phänotypischen Merkmals nahe. Darüber hinaus codieren sie normalerweise für kontinuierliche Merkmale und nicht für diskrete Merkmale.
Bei der Identifizierung der QTL -Regionen ist es wichtig, einen bestimmten QTL -Bereich sequenzieren. Darüber hinaus finden Untersuchungen und Forschungen, die Sequenzdaten der gemeinsamen QTL -Regionen mit der Beteiligung der Bioinformatik fest. Wir nennen diese Technik QTL Mapping. Anschließend baut eine Datenbank mit den Sequenzen der QTL -Region auf.

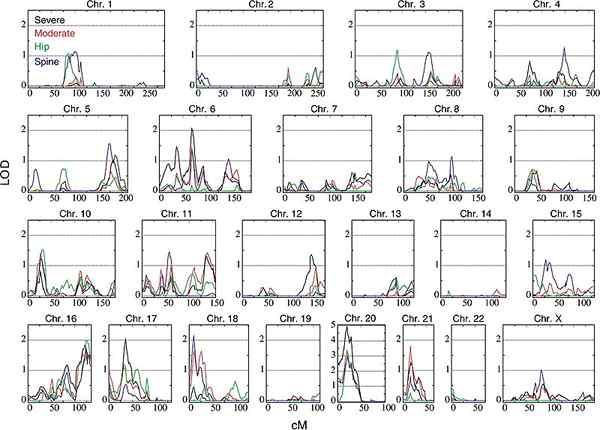
Abbildung 01: QTL -Scan
Darüber hinaus beruhen die Anwendungen von QTL -Sequenzen hauptsächlich auf BLAST -Tool, wobei die Sequenzähnlichkeit bestimmt werden kann. Es ist wichtig, die Beziehungen zwischen Organismen abzusetzen. Darüber hinaus ist es wichtig, die Komplexität eines bestimmten polygenen Phänotyps zu bestimmen. Es bestimmt auch die Entwicklung eines bestimmten Merkmals.
Derzeit wird die QTL -Analyse mit anderen Hochdurchsatztechniken wie DNA Microarrays kombiniert. Dies ist wichtig für die Expressionsanalyse des Phänotyps. Derzeit sind Wissenschaftler sehr an der Entwicklung der QTL -Datenbank interessiert, da QTL -Regionen für viele wichtige Merkmale verschiedener Arten verantwortlich sind.
Was ist GWAS?
GWAS steht für Genomweite Assoziationsstudie. Es bezieht sich auch auf Studien zur gesamten Genomassoziation. Diese Studien konzentrieren sich hauptsächlich auf Beobachtungsstudien. Es analysiert die genetischen Varianten verschiedener Individuen, die normalerweise mit einem bestimmten Merkmal assoziiert sind. Darüber hinaus ist das gesamte Genom für die GWAS -Analyse wichtig.
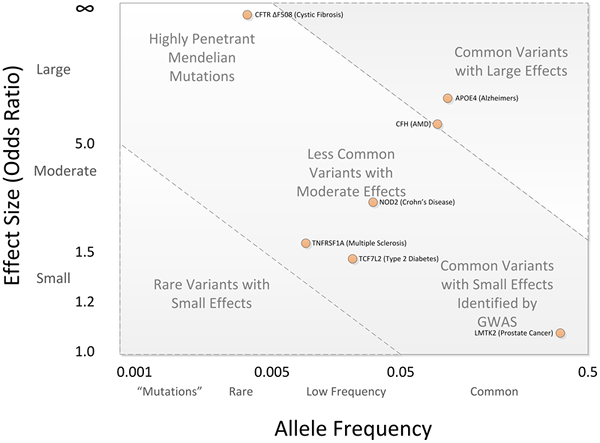
Abbildung 02: GWAS
GWAS ist ein wichtiges Instrument bei der Analyse von Einzelnukleotidpolymorphismen, die mit verschiedenen Erkrankungen verbunden sind. Dies ist auch eine vergleichende Untersuchung der verschiedenen einzelnen Nukleotidpolymorphismen über eine weite Population. Darüber hinaus ist die Studienprobe von GWAS sehr hoch; Daher nimmt es auch das Format einer Querschnittskohortenstudie an.
Darüber hinaus fand die erste GWAS -Studie im Hinblick auf den Myokardinfarkt und die Analyse der mit dem Myokardinfarkt verbundenen Gene statt. Derzeit spielt GWAS eine wichtige Rolle bei der Bestimmung des genetischen Hintergrunds komplexer Krankheiten mit unbekannter Ätiologie.
Was sind die Ähnlichkeiten zwischen QTL und GWAS?
- Sie stützen sich auf DNA -Sequenzen Daten für die Analyse.
- Beide sind mit den genetischen Hintergründen verschiedener Zeichen verbunden.
- Darüber hinaus benötigen sie Bioinformatik -Tools, um Ergebnisse abzuleiten und zu interpretieren.
- Sie sind auf großen Populationen durchgeführt.
Was ist der Unterschied zwischen QTL und GWAS?
QTL analysiert die phänotypischen Merkmale, die mit der polygenen Vererbung unter Verwendung verknüpfter Genloci assoziiert sind, während GWAS einzelne Nukleotidpolymorphismen einer bestimmten Erkrankung unter Verwendung des gesamten Genomsequenzen analysiert. Dies ist daher der Schlüsselunterschied zwischen QTL und GWAS. Die QTL -Mapping ist vergleichsweise eine weniger komplexe Technik als GWAS, da es eine gesamte Genomsequenzierung erfordert.
Die folgende Infografik fasst den Unterschied zwischen QTL und GWAS in tabellarischer Form zusammen.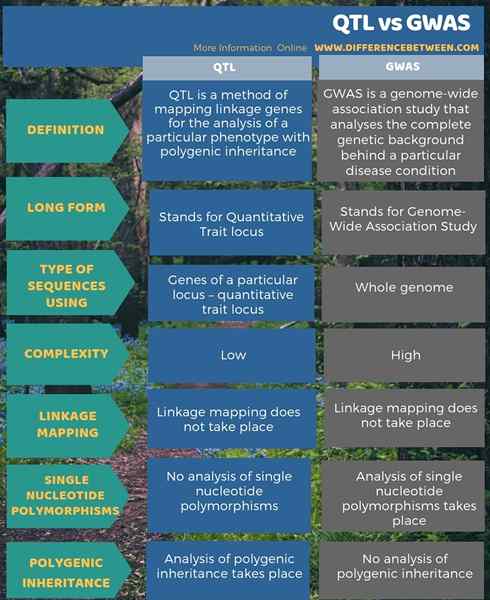
Zusammenfassung -Qtl gegen GWAS
QTL -Mapping und GWAS spielen eine wichtige Rolle bei der Bestimmung des genetischen Hintergrunds verschiedener phänotypischer Merkmale. Sie helfen auch bei der Analyse des genetischen Hintergrunds verschiedener Krankheiten. QTL -Mapping -Kartierung der Verknüpfungsgene, die für kontinuierliche Merkmale verantwortlich sind, und beinhaltet auch die Abbildung der polygenen Vererbungsgene. GWAS hingegen findet mit dem gesamten Genom statt, der auf Einzelnukleotidpolymorphismen analysiert wird. Darüber hinaus findet dies in einer größeren Bevölkerung statt. Dies ist daher die Zusammenfassung des Unterschieds zwischen QTL und GWAS.
Referenz:
1. Liu, Ruixian et al. „GWAS-Analyse und QTL-Identifizierung von Faserqualitätsmerkmalen und Ertragskomponenten in Hochlandbaumwolle unter Verwendung angereicherter SNP-Marker mit hoher Dichte.Grenzen in Pflanzenwissenschaften, Grenzen Medien s.A., 13. September. 2018, hier erhältlich.
2. „Gwas vs. Qtl Mapping?”Neueste Beiträge, die hier verfügbar sind.
Bild mit freundlicher Genehmigung:
1. „Beispiel für ein genomweit. (2003) Verknüpfung der Osteoporose mit Chromosom 20p12 und Assoziation mit BMP2. PLoS Biol 1 (3): E69 (CC durch 2.5) über Commons Wikimedia
2. "GWAS Disease Alleleffekte" von Bush WS und Moore JH - Bush WS, Moore JH (2012) Kapitel 11: Genomweite Assoziationsstudien. PLoS Comput Biol 8 (12): E1002822. doi: 10.1371/Journal.PCBI.1002822 (CC von 2.5) über Commons Wikimedia