Was ist der Unterschied zwischen ITS1 und ITS2
Der Schlüsselunterschied zwischen ITS1 und ITS2 ist, dass ITS1 eine Abstandshalter -DNA zwischen 18 und 5 ist.8S -rRNA -Gene in Eukaryoten, während ITS2 eine Spacer -DNA zwischen 5 ist.8S- und 28S -rRNA -Gene in Eukaryoten.
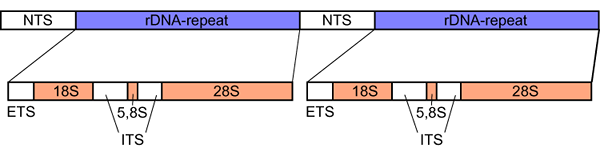
Interner transkribierter Spacer (IT) ist die Spacer -DNA, die sich normalerweise zwischen ribosomaler Ribosomal -Ribosomal -RNA -RNA -Genen der Untereinheit im Chromosom oder in der entsprechenden transkribierten Region im polyzistronischen rRNA -Vorläufermolekül befindet. In Bakterien und Archaea gibt es nur ein einzelnes IT zwischen 16 und 23 Jahren rRNA -Gene. Aber in Eukaryoten gibt es zwei seine Abstandshalter -DNA: ITS1 (zwischen 18 und 5).8S -rRNA -Gene) und ITS2 (zwischen 5.8s und 28s rRNA). Daher sind ITS1 und ITS2 zwei Abstandshalter -DNA, die zwischen rRNA -Genen in Eukaryoten gefunden wurden.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist seine 1
3. Was ist es2
4. Ähnlichkeiten - ITS1 und ITS2
5. Its1 gegen its2 in tabellarischer Form
6. Zusammenfassung
Was ist seine 1?
Its1 ist eine Abstandshalter -DNA zwischen 18 und 5 Jahren.8S -rRNA -Gene in Eukaryoten. Es bezieht sich auf ein Stück nicht funktionierender Abstandshalter -DNA zwischen rRNA -Genen. ITS1 hat eine größere Längenvariation als seine2. Die Längenvariation von ITS1 beträgt ungefähr (200-300 BP). Seine Marker wie ITS1 haben ihre Effizienz bei der Aufklärung der phylogenetischen Beziehungen zwischen verschiedenen Taxa bewiesen. Darüber hinaus ist seine 1 -Spacer -DNA weniger konserviert als seine 2 -Spacer -DNA. ITS1 -Strukturen werden nur in viel kleineren taxonomischen Einheiten konserviert. Jeder eukaryotische ribosomale Cluster enthält auch Regionen, die als externe transkribierte Abstandshalter bezeichnet werden (5 'und 3' ETs).

Abbildung 01: Its1 Region
Die ITS1 -Abstandshalter -DNA liegt sehr nahe am 5'externalen transkribierten Abstandshalter (5 'ETS). Wenn jemand die ITS1 -Region für Studien zur phylogenetischen Beziehungen amplifizieren möchte, müssen er ein bestimmtes Primerpaar (umgekehrte und vorwärts -Primer) für diesen Zweck verwenden. Um seine 1 -Region zu verstärken, werden normalerweise zwei Primer -Sets in den PCR -Protokollen von Laboratorien verwendet. Sie sind seine 1 vorwärts und seine 2 -umgekehrte oder sein 1 Foward und seine 4 -Rückseite.
Was ist es2?
Its2 ist eine DA -DNA der Abstandshalter, die sich zwischen 5 befindet.8S- und 28S -rRNA -Gene in Eukaryoten. Its2 Region hat eine kürzere Länge -Variation als die ITS1 -Region. Die Längenvariation von ITS2 ist ungefähr (180-240 bp). Die ITS2 -Region ist jedoch in vielen taxonomischen Einheiten konservierter als ihre 1 -Region. Es wird festgestellt, dass alle ITS2 -Sequenzen einen gemeinsamen Kern der Sekundärstruktur teilen. Darüber hinaus ist 40% der 2 -Region unter allen Angiospermen konserviert, während es möglich ist, 50% der ITS2 -Region über die Familie und höhere Ebene auszurichten.
Abbildung 02: Its2 Region
Unabhängig von dem Umfang der Erhaltung der Erhaltungsumfang der Verwendung der IS2 -Region, auch sehr beliebt, wie beispielsweise ihre 1 -Region in phylogenetischen Beziehungsstudien, insbesondere in der Pilz -DNA -Barcodierung. Um seine2 -Region zu verstärken, wird in den PCR -Protokollen von Laboratorien im Allgemeinen ein spezifischer Primersatz verwendet. Das ist es ist es 1 foward und its4 umgekehrt. Wenn das PCR -Protokoll diesen Primer -Set verwendet, verstärkt es auch seine 1 -Region zusammen mit der ITS2 -Region. Darüber hinaus liegt die ITS2 -Spacer -DNA sehr nahe am 3'externalen transkribierten Spacer (3 'ETS).
Ähnlichkeiten zwischen ITS1 und ITS2
- ITS1 und ITS2 sind zwei Spacer -DNA zwischen rRNA -Genen.
- Sie sind nur in Eukaryoten vorhanden.
- Beide können durch Verwendung spezifischer Primerpaare amplifiziert werden.
- Sie werden beide bei der rRNA-Reifung als nicht funktionale Nebenprodukte herausgeschnitten.
- Beide sind sehr wichtig für phylogenetische Beziehungsstudien, insbesondere bei Pilz -DNA -Barcodierung.
Unterschied zwischen ITS1 und ITS2
ITS1 ist eine Abstandshalter -DNA, die zwischen 18 und 5 vorhanden ist.8S -rRNA -Gene in Eukaryoten, während ITS2 eine Spacer -DNA zwischen 5 ist.8S- und 28S -rRNA -Gene in Eukaryoten. Dies ist also der Hauptunterschied zwischen ITS1 und ITS2. Darüber hinaus weist seine 1 eine größere Länge -Variation im Vergleich zu ITS2 auf. Andererseits hat ITS2 eine kürzere Längenschwankung im Vergleich zu ITS1. Dies ist ein weiterer Unterschied zwischen ITS1 und ITS2.
Die folgende Infografik listet die Unterschiede zwischen ITS1 und ITS2 in tabellarischer Form für Seite für Seitenvergleich auf.
Zusammenfassung -ITS1 gegen ITS2
Die interne transkribierte Region (IT) ist ein nicht funktionsfähiges Stück Spacer-DNA zwischen rRNA-Genen. In Bakterien und Archaea gibt es nur einen einzigen seiner Region. Aber in Eukaryoten gibt es zwei seine Regionen: ITS1 und ITS2. Its1 ist eine Abstandshalter -DNA, die sich zwischen 18 und 5 befindet.8S -rRNA -Gene in Eukaryoten, während ITS2 eine Spacer -DNA ist, die sich zwischen 5 befindet.8S- und 28S -rRNA -Gene in Eukaryoten. Dies ist also die Zusammenfassung des Unterschieds zwischen ITS1 und ITS2.
Referenz:
1. „Interner transkribierter Abstandhalter.”Ein Überblick | Sciencedirect -Themen.
2. Schoch, Conrad l., et al. „Kernribosomale interne transkribierte Spacer -Region (ITS) als universelle DNA -Barcode -Marker für Pilze.PNAs, Nationale Akademie der Wissenschaften.
Bild mit freundlicher Genehmigung:
1. "Eucaryot rdna" von - Pharaoh Han angenommen - keine maschinenlesbare Quelle zur Verfügung gestellt. Eigene Arbeit angenommen (basierend auf Urheberrechtsansprüchen) (CC BY-SA 3.0) über Commons Wikimedia
2. "Molekularmarkerssymbiodinium" von Allisonmlewis - eigene Arbeit (CC BY -SA 4.0) über Commons Wikimedia