Unterschied zwischen Exom- und RNA -Sequenzierung
Schlüsselunterschied - Exom gegen RNA -Sequenzierung
Die Nukleinsäure -Sequenzierung ist die Technik, die die Reihenfolge von Nukleotiden in einem bestimmten Fragment von DNA oder RNA eines Organismus bestimmt. Die Sequenzierung ist wichtig, um die DNA- und RNA -Make -up der Zelle zu identifizieren und bestimmte Gene zu unterscheiden, die für funktionelle Proteine codieren; Somit kann Sequenzierung verwendet werden, um die Mutationen dieser Gene und Genexpressionen zu verstehen. Die Sanger -Sequenzierungsmethode oder die fortgeschritteneren Sequenzierungsmethoden der nächsten Generation sind die häufig verwendeten Sequenzierungsmethoden. Die Exomsequenzierung ist die Sequenzierung des vollständigen Satzes von Exons oder kodierenden DNA -Regionen, die in einem Organismus vorhanden sind, während die RNA -Sequenzierung das Sequenzierungsverfahren von Ribonukleinsäuren (RNA) ist. Dies ist der Schlüsselunterschied zwischen Exom- und RNA -Sequenzierung.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist Exomsequenzierung
3. Was ist RNA -Sequenzierung
4. Ähnlichkeiten zwischen Exom- und RNA -Sequenzierung
5. Seite für Seitenvergleich - Exom vs RNA -Sequenzierung in tabellarischer Form
6. Zusammenfassung
Was ist Exomsequenzierung?
Exom ist eine Untergruppe des Genoms, das aus den kodierenden Genen eines bestimmten Organismus besteht. Die kodierenden Gene werden als Exons bezeichnet und in mRNA transkribiert und dann in Aminosäuresequenzen übersetzt. Während der Post -Transkriptions -Modifikationen entfernen der RNA -Spleißmechanismus in Eukaryoten die Introns (Nicht -Codierungsregionen), und die Exons bleiben bestehen. Es gibt zwei Haupttechniken, bei denen die Exomsequenzierung durchgeführt wird: lösungsbasiert und Arraybasiert.
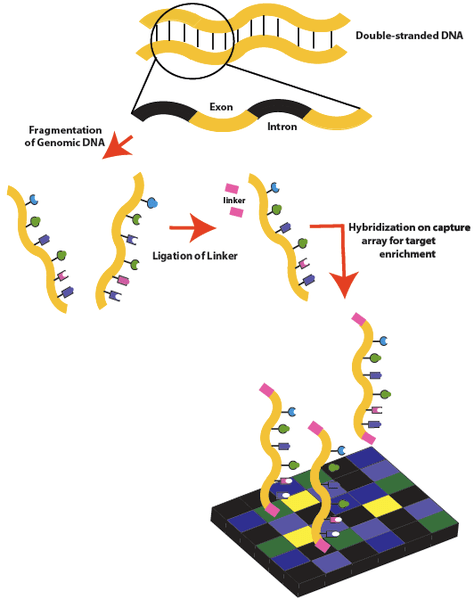
In lösungsbasierter Exomsequenzierung werden DNA-Proben unter Verwendung von Restriktionsenzymen oder mechanischer Methode fragmentiert und durch Wärme denaturiert. In dieser Technik werden biotinylierte Oligonukleotidsonden (Köder) verwendet, um mit Zielregionen im Genom selektiv zu hybridisieren. Magnetische Streptavidin -Perlen werden für den Bindungsschritt verwendet. Nach der Bindung folgt ein Waschschritt, bei dem die ungebundenen und nicht zielgerichteten Sequenzen weggespült werden. Die gebundenen Ziele werden dann unter Verwendung der Polymerasekettenreaktion (PCR) amplifiziert und dann unter Verwendung von Sanger -Sequenzierung oder Sequenzierungstechniken der nächsten Generation sequenziert.

Abbildung 01: Exomsequenzierung
Array -basierte Methode ähnelt auch der lösungsbasierten Methode, mit der Ausnahme, dass DNA.
Die Exomsequenzierung wird in vielen Anwendungen wie genetischer Diagnose von Krankheiten, in der Gentherapie bei der Identifizierung neuer genetischer Marker, in der Landwirtschaft verwendet.
Was ist RNA -Sequenzierung?
Die RNA -Sequenzierung basiert auf dem Transkriptom, bei dem es sich um die vollständigen Transkripte der Zelle handelt. Die wichtigsten Ziele der RNA-Sequenzierung sind es, alle Arten des Transkripts, einschließlich mRNA, nicht-kodierender RNA und kleiner RNA, zu katalogisieren, um die Transkriptionsstruktur von Genen zu bestimmen und die Expressionsniveaus jedes Transkripts während der Entwicklung zu quantifizieren. Während der RNA -Sequenzierung wurden zunächst Hybridisierungstechnologien (die komplementäre DNA aus reifen mRNA -Sequenzen waren) zur Sequenzierung verwendet. Gegenwärtig wird eine genauere und fortgeschrittene Durch-Put-Technik für die RNA-Sequenzierung verwendet.
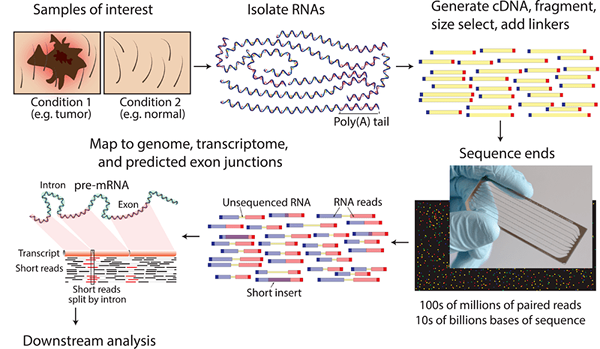
Abbildung 02: RNA -Sequenzierung
In der RNA -Sequenzierung wird eine Probe von RNA, die Gesamt -RNA oder fraktionierte RNA sein kann. Jedes cDNA -Fragment ist an Adaptern auf beiden Seiten (Paarendsequenzierung) oder auf einer Seite (Einzel -End -Sequenzierung) gebunden. Diese markierten Sequenzen werden unter Verwendung der Sanger -Sequenzierung oder der nächsten Generation wie der Exomsequenzierung sequenziert.
Was sind die Ähnlichkeiten zwischen Exom- und RNA -Sequenzierung?
- Kurz ausgewählte Fragmente oder der gesamte Satz von DNA / RNA können für die Exom- oder RNA -Sequenzierung verwendet werden.
- Sequenzierte Fragmente werden in Bibliotheken erhalten.
- Die Sanger -Sequenzierung oder Sequenzierung der nächsten Generation kann verwendet werden.
- Beide sind In -vitro -Sequenzierungsmethoden.
- Sequenzierte Fragmente können durch Fluoreszenz -Tags bestimmt werden.
Was ist der Unterschied zwischen Exom- und RNA -Sequenzierung?
Exom gegen RNA -Sequenzierung | |
| Die Exomsequenzierung ist die Sequenzierung des vollständigen Satzes von Exons oder codierenden DNA -Regionen, die in einem Organismus vorhanden sind. | Die RNA -Sequenzierung bezieht sich auf das Sequenzierungsverfahren von Ribonukleinsäuren (RNA); das Transkriptom. |
| Probe starten | |
| Genomische DNA ist die Startprobe der Exomsequenzierung. | RNA ist die Startprobe der RNA -Sequenzierung. |
| Komposition | |
| Dies enthält nur kodierende Regionen der gesamten DNA, die als Exons bekannt ist | Dies enthält RNA-mRNA / Transkriptom. |
| Sequenzierung | |
| Es gibt zwei Hauptmethoden zur Exomsequenzierung; Lösungsbasierte und Array -basierte Technologien. | Die RNA -Sequenzierung erfolgt durch die Herstellung einer cDNA -Bibliothek durch Extrahieren der gesamten RNA oder der fragmentierten RNA. |
Zusammenfassung -Exom gegen RNA -Sequenzierung
Exom ist der vollständige Satz von Codierungsregionen eines Organismus, und die Techniken, die an der Bestimmung der exakten Nukleotidordnung des Exoms beteiligt sind, werden als Exomsequenzierung bezeichnet. RNA -Sequenzierung ist die Technik, die an der Bestimmung der Nukleotidordnung der RNA eines Organismus beteiligt ist. Dies ist der Unterschied zwischen Exom- und RNA -Sequenzierung.
Laden Sie die PDF -Version von Exome vs RNA -Sequenzierung herunter
Sie können die PDF -Version dieses Artikels herunterladen und ihn für Offline -Zwecke gemäß Citation Note verwenden. Bitte laden Sie die PDF -Version hier den Unterschied zwischen Exom- und RNA -Sequenzierung herunter
Verweise:
1.Wang, Zhong et al. „RNA-Seq: Ein revolutionäres Instrument für die Transkriptomik.Naturbewertungen. Genetik, u.S. Nationalbibliothek für Medizin, Januar. 2009, hier erhältlich. Zugriff am 3. September. 2017.
2.Warr, Amanda et al. „Exome -Sequenzierung: aktuelle und zukünftige Perspektiven.G3: Gene | Genome | Genetik, Genetics Society of America, August. 2015, hier erhältlich. Zugriff am 3. September. 2017.
Bild mit freundlicher Genehmigung:
1. "Exome Sequencing Workflow 1a" von Malachi Griffith, Jason R. Walker, Nicholas C. Spione, Benjamin J. Ainscough, obi l. Griffith - (CC von 2.5) über Commons Wikimedia
2. "Tagebuch.PCBI.1004393.G002 ”von Sarahkusala - eigene Arbeit (CC von 3.0) über Commons Wikimedia