Unterschied zwischen Microarray und RNA -Sequenzierung
Schlüsselunterschied - Microarray gegen RNA -Sequenzierung
Transkriptom repräsentiert den gesamten Gehalt der in einer Zelle vorhandenen RNA, einschließlich mRNA, rRNA, tRNA, abgebautes RNA und nicht abgebauer RNA. Das Profiling -Transkriptom ist ein wichtiger Prozess, um die Zelleinsichten zu verstehen. Es gibt mehrere erweiterte Methoden für die Transkriptomprofilerstellung. Microarray- und RNA -Sequenzierung sind zwei Arten von Technologien, die zur Analyse von Transkriptom entwickelt wurden. Der Schlüsselunterschied zwischen Microarray und RNA -Sequenzierung ist das Microarray basiert auf dem Hybridisierungspotential von vorgegebenen markierten Sonden mit Ziel -cDNA -Sequenzen, während die RNA -Sequenzierung auf der direkten Sequenzierung von cDNA. Microarray wird mit dem Vorwissen über die Sequenzen durchgeführt und die RNA -Sequenzierung wird ohne das Vorkenntnis über Sequenzen durchgeführt.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist Microarray
3. Was ist RNA -Sequenzierung
4. Seite an Seitenvergleich - Microarray gegen RNA -Sequenzierung
5. Zusammenfassung
Was ist Microarray?
Microarray ist eine robuste, zuverlässige und hohe Durchsatzmethode, die für die Transkriptomprofilierung von Wissenschaftlern verwendet wird. Es ist der beliebteste Ansatz für die Transkriptanalyse. Es ist eine kostengünstige Methode, die von den Hybridisierungssonden abhängt.
Die Technik beginnt mit der Extraktion von mRNA aus der Probe und mit dem Bau der cDNA -Bibliothek aus der gesamten RNA. Dann wird es mit fluoreszenz markierten vorgefertigten Sonden auf einer festen Oberfläche gemischt (Spot -Matrix). Komplementäre Sequenzen hybridisieren mit den markierten Sonden im Microarray. Dann wird Microarray gewaschen und gescreent, und das Bild wird quantifiziert. Sammelte Daten sollten analysiert werden, um die relativen Expressionsprofile zu erhalten.
Es wird angenommen, dass die Intensität der Microarray -Sonden proportional zur Menge der Transkripte in der Probe ist. Die Genauigkeit der Technik hängt jedoch von den entworfenen Sonden, der Vorkenntnis der Sequenz und der Affinität der Sonden zur Hybridisierung ab. Daher hat die Microarray -Technologie Einschränkungen. Die Microarray -Technik kann nicht mit Transkripten mit geringer Häufigkeit durchgeführt werden. Es kann keine Isoformen differenzieren und genetische Varianten identifizieren. Da diese Methode von der Hybridisierung von Sonden, einigen Problemen im Zusammenhang mit der Hybridisierung wie Kreuzhybridisierung, nicht spezifischer Hybridisierung usw. abhängt, usw. tritt in der Microarray -Technik auf.

Abbildung 01: Microarray
Was ist RNA -Sequenzierung?
RNA -Schrotflintensequenzierung (RNA Seq) ist eine kürzlich entwickelte Gesamttranskriptom -Sequenzierungstechnik. Es ist eine schnelle und hohe Durchsatzmethode zur Transkriptomprofilierung. Es quantifiziert die Expression von Genen direkt und führt zu einer tiefen Untersuchung des Transkriptoms. RNA SEQ hängt nicht von vorgefertigten Sonden oder Vorkenntnis der Sequenzen ab. Daher hat die RNA -SEQ -Methode eine hohe Empfindlichkeit und Fähigkeit, neue Gene und genetische Varianten nachzuweisen.
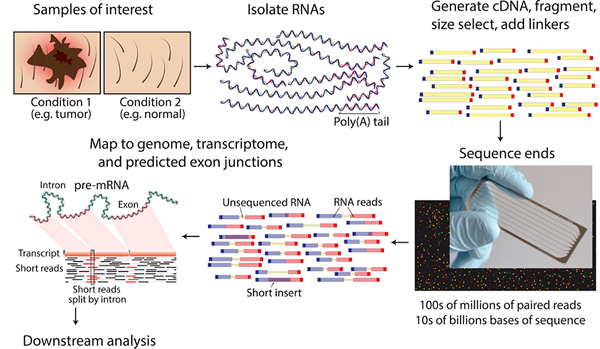
Die RNA -Sequenzierungsmethode wird über mehrere Schritte durchgeführt. Die Gesamt -RNA der Zelle muss isoliert und fragmentiert werden. Mithilfe der Reverse Transcriptase muss dann eine cDNA -Bibliothek erstellt werden. Jeder cDNA -Strang muss mit Adaptern ligiert werden. Dann müssen die ligierten Fragmente verstärkt und gereinigt werden. Mit einer NGS -Methode muss schließlich die Sequenzierung der cDNA durchgeführt werden.

Abbildung 02: RNA -Sequenzierung
Was ist der Unterschied zwischen Microarray und RNA -Sequenzierung?
Microarray gegen RNA -Sequenzierung | |
| Microarray ist eine robuste, zuverlässige Methode mit hohem Durchsatz. | Die RNA-Sequenzierung ist eine genaue und hohe Durchsatzmethode. |
| Kosten | |
| Dies ist eine kostengünstige Methode. | Dies ist eine teure Methode. |
| Analyse einer großen Anzahl von Proben | |
| Dies erleichtert die Analyse einer großen Anzahl von Proben gleichzeitig. | Dies erleichtert die Analyse einer großen Anzahl von Proben. |
| Datenanalyse | |
| Die Datenanalyse ist komplex. | In dieser Methode werden weitere Daten generiert. Daher ist der Prozess komplexer. |
| Vorkenntnis der Sequenzen | |
| Diese Methode basiert auf Hybridisierungssonden, daher Vorkenntnisse der Sequenzen in den erforderlichen Sequenzen. | Diese Methode hängt nicht vom vorherigen Sequenzwissen ab. |
| Strukturvariationen und neuartige Gene | |
| Diese Methode kann keine strukturellen Variationen und neuartigen Gene erkennen. | Diese Methode kann strukturelle Variationen wie Genverschmelzung, alternatives Spleißen und neue Gene erkennen. |
| Empfindlichkeit | |
| Dies kann keine Unterschiede in der Expression von Isoformen erkennen, daher hat dies eine begrenzte Empfindlichkeit. | Dies hat eine hohe Empfindlichkeit. |
| Ergebnis | |
| Dies kann nur zu relativen Expressionsniveaus führen. Dies ergibt keine absolute Quantifizierung der Genexpression. | Es gibt absolute und relative Expressionsniveaus. |
| Datenreanalyse | |
| Dies muss umgeleitet werden, um wieder zu analysieren. | Sequenzierungsdaten können erneut analysiert werden. |
| Bedarf an bestimmtem Personal und Infrastruktur | |
| Für Microarray sind keine spezifischen Infrastruktur und Personal erforderlich. | Spezifische Infrastruktur und Personal, die durch RNA -Sequenzierung erforderlich ist. |
| Technische Probleme | |
| Die Microarray-Technik hat technische Probleme wie Kreuzhybridisierung, unspezifische Hybridisierung, begrenzte Nachweisrate einzelner Sonden usw. | Die RNA-SEQ-Technik vermeidet technische Probleme wie Kreuzhybridisierung, unspezifische Hybridisierung, begrenzte Nachweisrate einzelner Sonden usw. |
| Vorurteile | |
| Dies ist eine voreingenommene Methode, da sie von der Hybridisierung abhängt. | Die Verzerrung ist im Vergleich zu Microarray niedrig. |
Zusammenfassung -Microarray gegen RNA -Sequenzierung
Microarray- und RNA -Sequenzierungsmethoden sind Plattformen mit hoher Durchsatz, die für die Transkriptomprofilierung entwickelt wurden. Beide Methoden erzeugen Ergebnisse, die stark mit Genexpressionsprofilen korrelieren. Die RNA -Sequenzierung hat jedoch Vorteile gegenüber Microarray für die Genexpressionsanalyse. Die RNA -Sequenzierung ist eine empfindlichere Methode zum Nachweis von Transkripten mit geringer Häufigkeit als Microarray. Die RNA -Sequenzierung ermöglicht auch die Differenzierung zwischen Isoformen und Identifizierung von Genvarianten. Microarray ist jedoch die gemeinsame Wahl der meisten Forscher, da die RNA -Sequenzierung eine neue und teure Technik mit Datenspeicherproblemen und komplexen Datenanalysen ist.
Verweise:
1.Wang, Zhong, Mark Gerstein und Michael Snyder. „RNA-Seq: Ein revolutionäres Instrument für die Transkriptomik.Naturbewertungen. Genetik. U.S. Nationalbibliothek für Medizin, Januar. 2009. Netz. 14. März. 2017
2.Rogler, Charles E., Tatyana Tchaikovskaya, Raquel Norel, Aldo Massimi, Christopher Plescia, Eugeny Rubashevsky, Paul Siebert und Leslie E. Rogler. „RNA-Expression Microarrays (REMS), eine Hochdurchsatzmethode zur Messung der Unterschiede in der Genexpression in verschiedenen biologischen Proben.”Nukleinsäuren erforschen. Oxford University Press, 01. Januar. 2004. Netz. 15. März. 2017
3.Zhao, Shanrong, Wai-Ping Pilg-Leung, Anton Bittner, Karen Ngo und Xuejun Liu. „Vergleich von RNA-Seq und Microarray in der Transkriptomprofilierung aktivierter T-Zellen." PLUS EINS. Öffentliche Wissenschaftsbibliothek, Januar. 2014. Netz. 15. März. 2017
Bild mit freundlicher Genehmigung:
1. "Tagebuch.PCBI.1004393.G002 ”von Malachi Griffith, Jason R. Walker, Nicholas C. Spione, Benjamin J. Ainscough, obi l. Griffith - (CC von 2.5) über Commons Wikimedia
2. "Microarray" von Bill Branson (Fotograf) - National Cancer Institute (Public Domain) über Commons Wikimedia