Unterschied zwischen NGS und Sanger -Sequenzierung
Schlüsselunterschied - NGS vs Sanger -Sequenzierung
Sequenzierung der nächsten Generation (NGS) und Sanger -Sequenzierung sind zwei Arten von Nucleotid -Sequenzierungstechniken, die im Laufe der Zeit entwickelt wurden. Die Sanger -Sequenzierungsmethode wurde seit vielen Jahren weit verbreitet und NGS ersetzt sie kürzlich aufgrund ihrer Vorteile. Der Schlüsselunterschied zwischen NGS und Sanger -Sequenzierung ist das NGS funktioniert auf das Prinzip der Sequenzierung von Millionen von Sequenzen gleichzeitig schnell durch ein Sequenzierungssystem Elektrophorese.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist die Nukleotid -Sequenzierung
3. Was ist NGS
4. Was ist Sanger -Sequenzierung
5. Seite an Seitenvergleich - NGS vs Sanger -Sequenzierung
6. Zusammenfassung
Was ist die Nukleotid -Sequenzierung?
Genetische Informationen werden in den Nukleotidsequenzen der DNA oder RNA eines Organismus gespeichert. Der Prozess der Bestimmung der korrekten Reihenfolge von Nukleotiden (unter Verwendung von vier Basen) in einem gegebenen Fragment (in einem Gen, einer Cluster von Genen, Chromosom und vollständigem Genom) wird als Nukleotidsequenzierung bezeichnet. In genomischen Studien, forensischen Studien, Virologie, biologischer systematischer, medizinischer Diagnose, Biotechnologie und vielen anderen Bereichen ist es sehr wichtig, die Struktur und Funktion von Genen zu analysieren. Es gibt verschiedene Arten von Sequenzierungsmethoden, die von Wissenschaftlern entwickelt wurden. Darunter, Sanger -Sequenzierung Der 1977 von Frederick Sanger entwickelte von Frederick Sanger wurde weit verbreitet und populär Sequenzierung der nächsten Generation ersetzt es.
Was ist NGS?
Die Sequenzierung der nächsten Generation (NGS) ist ein Begriff. Es beschreibt eine Reihe verschiedener moderner Sequenzierungstechnologien, die genomische Studien und molekulare Biologie revolutionierten. Diese Techniken sind Illumina -Sequenzierung, Roche 454 -Sequenzierung, Ionenprotonsequenzierung und feste Sequenzierung (Sequenzierung durch Oligo -Ligation). NGS -Systeme sind schneller und billiger. In NGS -Systemen werden vier HauptdNA -Sequenzierungsmethoden verwendet; Pyrosequenzierung, Sequenzierung durch Synthese, Sequenzierung durch Ligation und Ionen -Halbleitersequenzierung. Eine große Anzahl von DNA- oder RNA -Strängen (Millionen von) kann parallelly sequenziert werden. Es ermöglicht die Sequenzierung des gesamten Genoms von Organismen innerhalb eines kurzen Zeitraums, im Gegensatz zu Sanger -Sequenzierung, die mehr Zeit in Anspruch nehmen.
NGS hat viele Vorteile gegenüber der konventionellen Sequenzierung Sanger -Methode. Es ist ein hoher Geschwindigkeits-, genauerer und kostengünstigerer Prozess, der mit einer kleinen Stichprobengröße durchgeführt werden kann. NGs können in metagenomischen Studien verwendet werden, um Variationen innerhalb eines einzelnen Genoms aufgrund von Insertionen und Deletionen usw. zu erkennen usw. und bei der Analyse von Genausdrücken.
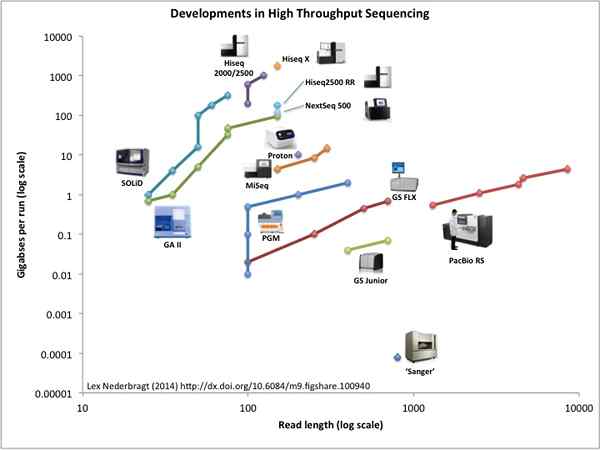
Abbildung_1: Entwicklungen in der NGS -Sequenzierung
Was ist Sanger -Sequenzierung?
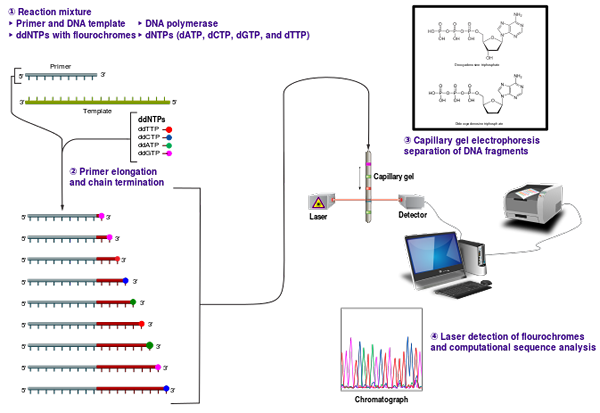
Die Sanger -Sequenzierung ist eine Sequenzierungsmethode, die 1977 von Frederick Sanger und seinen Kollegen entwickelt wurde, um die genaue Nukleotidordnung eines bestimmten DNA -Fragments zu bestimmen. Es ist auch als bekannt als Kettenabschlussequenzierung oder Dideoxy -Sequenzierung. Das Arbeitsprinzip dieser Methode ist die Beendigung der Strangsynthese durch selektives Einbau einer Kette, die Dideoxynukleotide (DDNTPs) wie DDGTP, DDCTP, DDATP und DDTTP durch DNA -Polymerase während der Replikation von DNA. Normale Nukleotide haben 3 'OH -Gruppen zur Bildung einer Phosphodiesterbindung zwischen benachbarten Nukleotiden, um die Strangbildung fortzusetzen. DDNTPs fehlt jedoch in dieser 3' -OH -Gruppe und können keine Phosphodiesterbindungen zwischen Nukleotiden bilden. Daher wird die Kettenverlängerung eingestellt.
Bei dieser Methode dient die zu sequenzierte einsträngige DNA als Vorlagenstrang für in vitro DNA -Synthese. Weitere Anforderungen sind Oligonukleotidprimer, Desoxynukleotid -Vorläufer und DNA -Polymerase -Enzym. Wenn die flankierenden Enden des Zielfragments bekannt sind, können Primer leicht für die DNA -Replikation ausgelegt werden. In vier separaten Röhrchen werden vier separate DNA -Synthesereaktionen durchgeführt. Jedes Röhrchen hat separate DDNTPs zusammen mit anderen Anforderungen. Aus dem jeweiligen Nukleotid werden eine Mischung aus DNTPs und DDNTPs zugesetzt. Ebenso werden vier separate Reaktionen in vier Röhrchen mit vier Gemischen durchgeführt. Nach den Reaktionen wird der Nachweis von DNA -Fragmenten und die Umwandlung des Fragmentmusters in Sequenzinformationen durchgeführt. Die resultierenden DNA -Fragmente werden wärme -denaturiert und durch Gelelektrophorese getrennt. Wenn radioaktive Nukleotide verwendet werden, kann das Bändermuster im Polyacrylamidgel durch Autoradiographie sichtbar gemacht werden. Wenn diese Methode die fluoreszenz markierten Dideoxynukleotide verwendet, kann sie im gelesenen Gel gemindert und durch einen Laserstrahl geleitet werden, der vom Fluoreszenzdetektor nachgewiesen werden soll. Um Fehler zu vermeiden, die auftreten könnten, wenn eine Sequenz vom Auge gelesen wird und manuell in einen Computer eintritt.
Dies ist die Methode, mit der DNA vom menschlichen Genomprojekt sequenziert wurde. Diese Methode wird immer noch mit erweiterten Änderungen verwendet, da sie genaue Sequenzinformationen enthält, obwohl sie ein teurer und langsamer Prozess sind.

Abbildung_2: Sanger -Sequenzierung
Was ist der Unterschied zwischen NGS und Sanger -Sequenzierung?
NGS gegen Sanger -Sequenzierung | |
| Sequenzierung der nächsten Generation (NGS) beziehen sich auf moderne Sequenzierungsprozesse mit hohem Durchsatz. Es beschreibt eine Reihe verschiedener moderner Sequenzierungstechnologien | Die Sanger -Sequenzierung ist eine Sequenzierungsmethode, die von Frederick Sanger entwickelt wurde, um die genaue Nukleotidordnung eines gegebenen DNA -Fragments zu bestimmen. |
| Kosteneffektivität | |
| NGS ist ein billigerer Prozess, weil es Zeit, Menschenleistung und Chemikalien reduziert. | Dies ist ein kostspieliger Prozess, da es Zeit, Menschenleistung und mehr Chemikalien braucht. |
| Geschwindigkeit | |
| Dies ist schneller, da sowohl chemische Erkennung als auch Signalerkennung vieler Stränge parallel stattfinden. | Dies ist zeitaufwändig, da der chemische Erkennung und die Signalerkennung als zwei getrennte Prozesse und nur auf Strand gleichzeitig lesen können. |
| Zuverlässigkeit | |
| NGS ist zuverlässig. | Die Sanger -Sequenzierung ist weniger zuverlässig |
| Probengröße | |
| NGs erfordert weniger DNA -Menge an DNA. | Diese Methode benötigt eine große Menge an Vorlagen -DNA. |
| DNA -Basen pro sequenziertes Fragment | |
| Die Anzahl der DNA -Basen pro sequenziertes Fragment ist niedriger als die Sanger -Methode | Erzeugungsequenzen sind länger als NGS -Sequenzen. |
Zusammenfassung - NGS vs Sanger -Sequenzierung
NGS- und Sanger -Sequenzierung sind Nucleotid -Sequenzierungstechniken, die in der molekularen Biologie ausführlich verwendet werden. Die Sanger -Sequenzierung ist eine frühe Sequenzierungsmethode, die durch NGs ersetzt wurde. Der Hauptunterschied zwischen NGS und Sanger-Sequenzierung besteht darin, dass NGS ein hoher Geschwindigkeits-, genauerer und kostengünstigerer Prozess als Sanger-Sequenzierung ist. Beide Techniken haben wichtige Ausbrüche in Genetik und Biotechnologie erstellt.
Referenz:
1. Nowrousian, Minou. „Sequenzierungstechniken der nächsten Generation für eukaryotische Mikroorganismen: Sequenzierungsbasierte Lösungen für biologische Probleme." Eukaryotische Zelle. American Society for Microbiology, September. 2010. Netz. 18. Februar. 2017
2. Sanger, f., S. Nicklen und a. R. Coulson. „DNA-Sequenzierung mit Ketten-terminierenden Inhibitoren.Verfahren der Nationalen Akademie der Wissenschaften 74.12 (1977): 5463-467. Netz.
3. Liu, Lin, Yinhu Li, Siliang Li, ni Hu, Yimin HE, Ray Pong, Danni Lin, Lihua Lu und Maggie Law. „Vergleich von Sequenzierungssystemen der nächsten Generation.Journal of Biomedicine and Biotechnology 2012 (2012): 1-11. Netz.
Bild mit freundlicher Genehmigung:
"Sanger-Sequencing" von Estevezj-eigene Arbeit (CC BY-SA 3.0) über Commons Wikimedia
„Entwicklungen in der Sequenzierung der nächsten Generation“ von Nederbragt, Lex (2012) - (CC von 3.0) über Commons Wikimedia