Unterschied zwischen Sanger -Sequenzierung und Pyrosequenzierung
Schlüsselunterschied - Sanger -Sequenzierung im Vergleich zur Pyrosequenzierung
Die DNA -Sequenzierung ist für die DNA -Analyse sehr wichtig, da die Kenntnis der korrekten Nukleotideanordnung in einer bestimmten DNA -Region viele wichtige Informationen darüber zeigt. Es gibt verschiedene DNA -Sequenzierungsmethoden. Sanger -Sequenzierung und Pyrosequenzierung sind zwei verschiedene DNA -Sequenzierungsmethoden, die in der molekularen Biologie weit verbreitet sind. Der Schlüsselunterschied zwischen Sanger -Sequenzierung und Pyrosequenzierung besteht darin Die Sanger -Sequenzierung verwendet Dideoxynukleotide, um die Synthese von DNA zu beenden, um die Nucleotidsequenz zu lesen, während die Pyrosequenzierung die Pyrophosphatfreisetzung durch Einbau der Nukleotide und die Synthese der Komplementärsequenz nachlässt, um die genaue Reihenfolge der Sequenz zu lesen.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist Sanger -Sequenzierung
3. Was ist Pyrosequencing
4. Seite an Seitenvergleich - Sanger -Sequenzierung gegen Pyrosequenzierung
5. Zusammenfassung
Was ist Sanger -Sequenzierung?
Die Sanger -Sequenzierung ist eine DNA -Sequenzierungsmethode der ersten Generation, die von Frederick Sanger und seinen Colleges im Jahr 1977 entwickelt wurde. Es ist auch als bekannt als Kettenabschlussequenzierung oder Dideoxy -Sequenzierung Da es auf der Kettenbeendigung durch Dideoxynukleotide (ddntps) basiert, basiert es. Diese Methode wurde seit mehr als 30 Jahren weit verbreitet, bis die Sequenzierung der neuen Generation (NGS) entwickelt wurde. Die Sanger -Sequenzierungstechnik ermöglichte die Entdeckung der richtigen Nukleotidreihenfolge oder die Anhaftung eines bestimmten DNA -Fragments. Es basiert auf dem selektiven Einbau von DDNTPs und der Beendigung der DNA -Synthese während der in vitro DNA Replikation. Das Fehlen von 3' -OH -Gruppen, um die Bildung von Phosphodiesterbindungen zwischen benachbarten Nukleotiden fortzusetzen, ist ein einzigartiges Merkmal von DDNTPs. Sobald der DDNTP angeschlossen ist, hört die Kettenverlängerung ab und endet von diesem Punkt. Es gibt vier DDNTPs - DDATP, DDCTP, DDGTP und DDTTP - in der Sanger -Sequenzierung verwendet. Diese Nukleotide stoppen den DNA -Replikationsprozess, wenn sie in den wachsenden DNA -Strang eingebaut werden, und führen zu unterschiedlichen Längen der kurzen DNA. Kapillargelelektrophorese wird verwendet, um diese kurzen DNA -Stränge nach ihren Größen auf einem Gel zu organisieren, wie in Abbildung 01 gezeigt.
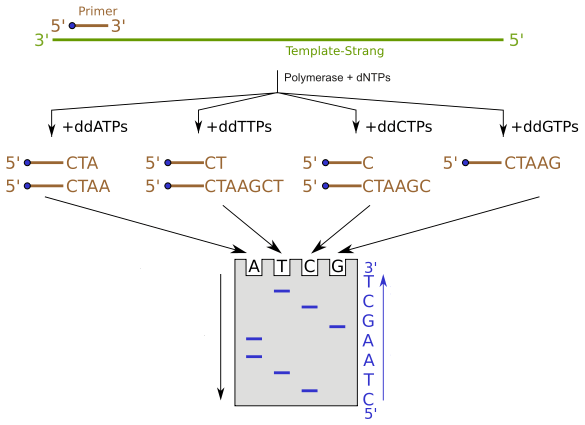
Abbildung 1: Kapillargelelektrophorese synthetisierter kurzer DNA
Für in vitro Replikation von DNA, nur wenige Anforderungen sollten erfüllt werden. Sie sind DNA -Polymerase -Enzym, Template -DNA, Oligonukleotidprimer und Desoxynukleotide (DNTPs). In der Sanger -Sequenzierung wird die DNA. Desoxynukleotide werden nicht vollständig durch die jeweiligen DDNTPs ersetzt. Eine Mischung aus dem jeweiligen DNTP (z. B. DATP + DDATP) ist im Röhrchen enthalten und repliziert. Vier separate Rohrprodukte werden in vier separaten Brunnen auf einem Gel ausgeführt. Dann kann durch das Lesen des Gels die Sequenz konstruiert werden, wie in Abbildung 02 gezeigt.

Abbildung 02: Sanger -Sequenzierung
Die Sanger -Sequenzierung ist eine wichtige Technik, die in vielen Bereichen der molekularen Biologie hilft. Das Human-Genom-Projekt wurde mit Hilfe von Sanger-Sequenzierungsmethoden erfolgreich abgeschlossen. Die Sanger -Sequenzierung ist auch nützlich für die Ziel -DNA -Sequenzierung, Krebs- und genetische Erkrankungen, die Genexpressionsanalyse, die Identifizierung des Menschen, die Erkennung von Pathogen, mikrobielle Sequenzierung usw.
Es gibt mehrere Nachteile der Sanger -Sequenzierung:
- Die Länge der sequenzierten DNA kann nicht länger als 1000 Basispaare betragen
- Es kann jeweils nur ein Strang sequenziert werden.
- Der Prozess ist zeitaufwändig und teuer.
Daher wurden neue fortschrittliche Sequenzierungstechniken entwickelt, um diese Probleme zu überwinden. Die Sanger -Sequenzierung wird jedoch immer noch verwendet.
Was ist Pyrosequencing?
Pyrosequencing ist eine neuartige DNA -Sequenzierungstechnik, die auf der „Sequenzierung durch Synthese“ basiert,. Diese Technik beruht auf dem Nachweis der Pyrophosphatfreisetzung beim Nucleotid -Einbau. Das Verfahren wird von vier verschiedenen Enzymen verwendet: DNA -Polymerse, ATP -Sulfurylase, Luciferase und Apyrase sowie zwei Substraten Adenosin 5 'Phosphosposulfat (APS) und Luciferin.
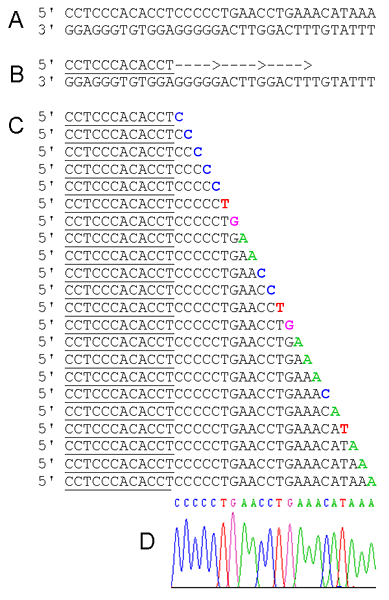
Der Prozess beginnt mit der Primerbindung mit der einzelsträngigen DNA-Template und DNA-Polymerase beginnt den Einbau von Nukleotiden. Wenn sich die Nukleotide miteinander verbinden (Nukleinsäurepolymerisation), setzt sie Pyrophosphat (zwei zusammengebundene Phosphatgruppen) und Energie frei. Jede Nukleotid -Addition fördert die äquimolare Menge an Pyrophosphat. Pyrophosphat konvertiert durch ATP -Sulfylylase in Gegenwart von Substrat -APs in ATP. Das erzeugte ATP treibt die Luciferase-vermittelte Umwandlung von Luciferin in Oxyluciferin an und produziert sichtbares Licht in Mengen, die proportional zur Menge an ATPs sind. Licht wird durch ein Photonenerkennungsgerät oder durch Fotomultiplikator erkannt und erstellt ein Pyrogramm. Apyrase beeinträchtigt ATP und nicht eingebaute DNTPs im Reaktionsgemisch. DNTP -Addition erfolgt einmal einmal. Da die Zugabe von Nukleotid gemäß dem Einbau und Nachweis von Licht bekannt ist, kann die Abfolge der Vorlage bestimmt werden. Pyrogramm wird zur Erzeugung der Nukleotidsequenz der Proben -DNA verwendet, wie in Abbildung 03 gezeigt.
Pyrosequenzierung ist bei der Analyse des einzelnen Nucleotidpolymorphismus und der Sequenzierung kurzer DNA sehr wichtig. Die hohe Genauigkeit, Flexibilität, einfache Automatisierung und parallele Verarbeitung sind die Vorteile der Pyrosequenzierung gegenüber Sanger -Sequenzierungstechniken.
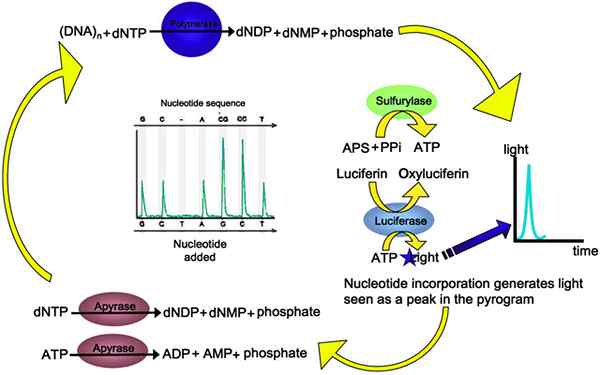
Abbildung 03: Pyrosequenzierung
Was ist der Unterschied zwischen Sanger -Sequenzierung und Pyrosequenzierung?
Sanger -Sequenzierung gegen Pyrosequenzierung | |
| Die Sanger -Sequenzierung ist eine DNA -Sequenzierungsmethode, die auf dem selektiven Einbau von DDNTPs durch DNA -Polymerase und Kettenabschluss basiert. | Pyrosequenzierung ist eine DNA -Sequenzierungsmethode, die auf dem Nachweis der Pyrophosphatfreisetzung beim Einbau von Nukleotid basiert. |
| Verwendung von ddntp | |
| DDNTPs werden verwendet, um die DNA -Replikation zu beenden | DDNTPs werden nicht verwendet. |
| Beteiligte Enzyme | |
| DNA -Polymerase werden verwendet. | Es werden vier Enzyme verwendet: DNA -Polymerase, ATP -Sulfurylase, Luciferase und Apyrase. |
| Substrate verwendet | |
| APs und Luciferin werden nicht verwendet. | Adenosin 5 'Phosphosulfat (APS) und Luciferin werden verwendet. |
| Maximale Temperatur | |
| Dies ist ein langsamer Prozess. | Dies ist ein schneller Prozess. |
Zusammenfassung -Sanger -Sequenzierung im Vergleich zur Pyrosequenzierung
Sanger -Sequenzierung und Pyrosequenzierung sind zwei DNA -Sequenzierungsmethoden, die in der molekularen Biologie verwendet werden. Die Sanger -Sequenzierung konstruiert die Reihenfolge der Nukleotide nach Abschluss der Kettenverlängerung, während die Pyrosequenzierung die genaue Reihenfolge der Nukleotide durch Einbau von Nukleotiden und Nachweis der Freisetzung von Pyrophosphaten konstruiert. Daher besteht der Hauptunterschied zwischen Sanger -Sequenzierung und Pyrosequenzierung darin, dass die Sanger -Sequenzierung durch Kettenabschlüsse funktioniert, während die Pyrosequenzierung durch Synthese zur Sequenzierung funktioniert.
Referenz:
1. Fakruddin, MD und Abhijit Chowdhury. „Pyrosequenzierung-eine Alternative zur traditionellen Sanger-Sequenzierung.American Journal of Biochemistry and Biotechnology. Wissenschaftspublikationen, 02. März. 2012. Netz. 28. Februar. 2017.
2. „Sanger -Sequenzierung.”Sanger -Sequenzierung - Sciencedirect -Themen. N.P., N.D. Netz. 28. Februar. 2017
Bild mit freundlicher Genehmigung:
1. "Didesoxy -Methode" von Christoph Goemans (Modifiziert) - Dr. Norman Mauder, auf Basis Einer Datei von Christoph Goemans (CC BY-SA 3.0) über Commons Wikimedia
2. "Sanger-DNA-seq" von Enzo bei der polnischen Sprachwikipedia (CC BY-SA 3.0) über Commons Wikimedia
3. "Pyrosequencing" von microbiologyBytes (CC BY-SA 2.0) über Flickr