Was ist der Unterschied zwischen CGH und Array CGH
Der Schlüsselunterschied Zwischen CGH und Array CGH ist, dass CGH eine traditionelle molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe mit einer geringeren Auflösung ist, während Array CGH eine vollständig automatisierte molekulare zytogenetische Methode zur Analyse der Kopienzahlvariation in DNA einer Testprobe ist, die a hat höhere Auflösung.
CGH und Array CGH sind zwei Techniken, um die Variationen der Kopienzahl bei Individuen zu erkennen. Die Variation der Kopienzahl bezieht sich auf die Abschnitte des Genoms, die wiederholt werden. Die Kopienzahl variiert zwischen Individuen. Die Variation der Kopienzahl wird im Allgemeinen in zwei Gruppen eingeteilt: kurze Wiederholungen und lange Wiederholungen. Eines der bekanntesten Beispiele für eine kurze Variation von Kopienzahlen ist die Wiederholung des CAG-Trinukleotids im Huntingtin-Gen. Dies führt zu einer neurologischen Störung namens Huntingtin. Das beste Beispiel für lange Wiederholungen Die Variation der Kopienzahl ist das Alpha -Amylase -Gen, das Insulinresistenz verursacht. Die Variation der Kopienzahl kann durch Techniken wie Fisch, CGH und Array CGH erkannt werden.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist CGH
3. Was ist Array CGH
4. Ähnlichkeiten - CGH und Array CGH
5. CGH gegen Array CGH in tabellarischer Form
6. Zusammenfassung - CGH gegen Array CGH
Was ist CGH?
CGH ist eine traditionelle molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe im Vergleich zu einer Referenzprobe. Es hat eine geringere Auflösung im Vergleich zu automatisierten Techniken. Das Ziel dieser Technik ist es, zwei genomische DNA -Proben schnell und effizient zu vergleichen, die aus verschiedenen Quellen entstehen.
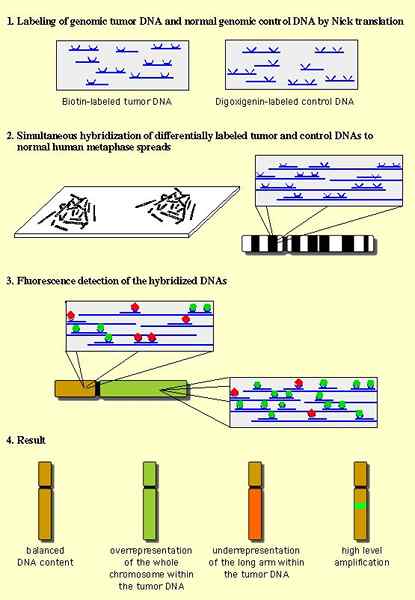
Abbildung 01: CGH
Das Verfahren umfasst die Isolierung von DNA aus zwei Quellen: Test und Referenz. Dann ist der nächste Schritt die unabhängige Markierung von Test- und Referenz -DNA -Proben mit Fluorophoren verschiedener Farben. Später wurden die beiden DNA-Proben denaturiert, um die einsträngigen DNA-Proben zu erhalten. Schließlich wurde die Hybridisierung der beiden resultierenden DNA -Proben in einem Verhältnis von 1: 1 zu einer normalen Metaphase -Verbreitung von Chromosomen durchgeführt. Die chromosomalen Unterschiede können zwischen den beiden Quellen unter Verwendung eines Fluoreszenzmikroskops und einer Computersoftware identifiziert werden. Darüber hinaus kann die CGH -Technik nur unausgeglichene chromosomale Anomalien nachweisen.
Was ist Array CGH?
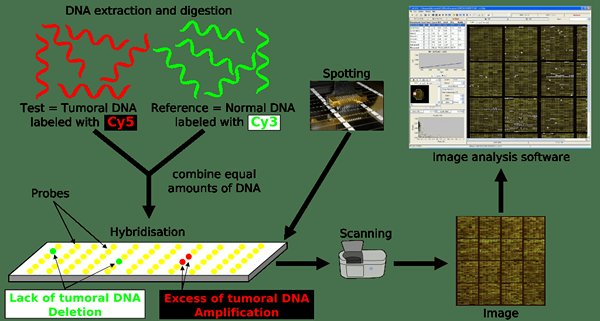
Array CGH ist eine vollständig automatisierte molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe im Vergleich zu einer Referenzprobe. Es hat eine höhere Auflösung und wurde entwickelt, um die Einschränkungen des traditionellen CGH zu minimieren. Array CGH kombiniert die Prinzipien von CGH unter Verwendung von Microarrays. Anstatt Metaphase -Chromosomen zu verwenden, wird diese Methode mit kleinen DNA -Segmenten als Ziele mit Objektträgern ausgestattet. Diese Microarrays wurden durch die Ablagerung und Immobilisierung einer kleinen Menge DNA (Sonden) auf einer festen Glasstütze hergestellt. Die Größe der Sonden kann von Oligonukleotid zu einem bakteriellen künstlichen Chromosom variieren.

Abbildung 02: Array CGH
Unabhängig von den Sonden ähnelt die grundlegende Methode des Array -CGH der CGH. Sobald Sonden, die Microarray enthalten. Da die DNA in den Proben aufgrund der Denaturierung einzelner Strandung ist. Mit einem digitalen Bildgebungssystem ist es möglich, die chromosomalen Unterschiede zwischen den beiden Quellen zu identifizieren.
Was sind die Ähnlichkeiten zwischen CGH und Array CGH?
- CGH und Array CGH sind zwei Techniken, um Kopienzahlvariationen zu erkennen.
- Sie sind molekulare zytogenetische Methoden.
- Beide Methoden diagnostizieren unausgeglichene chromosomale Anomalien.
- Fluorophore sind an beiden Methoden beteiligt.
- Beide Methoden können verwendet werden, um genetische Störungen aufgrund von Kopienzahlvariationen nachzuweisen.
- Sie werden in der vergleichenden genomischen Analyse häufig verwendet.
Was ist der Unterschied zwischen CGH und Array CGH?
CGH ist eine traditionelle molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe, die eine geringere Auflösung aufweist, während Array -CGH eine vollständig automatisierte molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe ist. Dies ist also der Hauptunterschied zwischen CGH und Array CGH. Darüber hinaus hybridisieren Sie in CGH-, Test- und Referenz -DNA -Proben mit einer normalen Metaphase -Verbreitung von Chromosomen.
Die folgende Infografik listet die Unterschiede zwischen CGH und Array CGH in tabellarischer Form für Seite für Seitenvergleich auf.
Zusammenfassung -CGH gegen Array CGH
CGH und Array CGH sind zwei molekulare zytogenetische Techniken, um unausgeglichene Chromosomanomalien nachzuweisen. Sie werden in der vergleichenden genomischen Analyse häufig verwendet. CGH ist eine traditionelle molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe, die eine niedrigere Auflösung aufweist. Im Gegensatz dazu ist Array CGH eine vollständig automatisierte molekulare zytogenetische Methode zur Analyse der Variation der Kopienzahl in der DNA einer Testprobe mit einer höheren Auflösung. Dies fasst den Unterschied zwischen CGH und Array CGH zusammen.
Referenz:
1. „Vergleichende genomische Hybridisierung.”Ein Überblick | Sciencedirect -Themen.
2. Heather, James M., und Benjamin -Kette. „Die Folge der Sequenzer: Die Geschichte der Sequenzierung der DNA.Genomik, akademische Presse, 10. November. 2015.
Bild mit freundlicher Genehmigung:
1. "CGH -Schema" von Mbaraudis (Talk) (Uploads) - eigene Arbeit (CC von 2.5) über Commons Wikimedia
2. "Array-CGH-Protokoll" von Philippe Hupé-Emmanuel Barillot, Laurence Calzone, Philippe Hupé, Jean-Philippe Vert, Andrei Zinovyev, Computersysteme Biologie des Krebs Chapman & „Hall/CRC Mathematical“ Computational Biology, 2012 (CC BY-SA 3 3.0) über Commons Wikimedia