Unterschied zwischen Nichtübereinstimmungreparatur und Nucleotid -Exzisionsreparatur
Schlüsselunterschied - Reparaturenfehlanpassungsreparatur gegenüber Nucleotid -Exzisionsreparaturen
Zehn- und Tausende von DNA -Schäden treten in der Zelle pro Tag auf. Es induziert Veränderungen der Zellprozesse wie Replikation, Transkription sowie der Lebensfähigkeit der Zelle. In einigen Fällen können Mutationen, die durch diese DNA-Schäden verursacht werden, zu schädlichen Krankheiten wie Krebs und alternassoziiertem Syndromen (z. B. Progerie) führen. Unabhängig von diesen Schäden initiiert die Zelle einen hoch organisierten Kaskadenreparaturmechanismus, der als DNA -Schadensreaktionen bezeichnet wird. Im zellulären System wurden mehrere DNA -Reparatursysteme identifiziert. Diese sind als Basis -Exzisionsreparatur (BER), Mismatchreparatur (MMR), Nucleotid Exzisionsreparatur (NER), Doppelstrang -Bruchreparatur bekannt. Die Reparatur von Nucleotid -Exzisionsreparaturen ist ein sehr vielseitiges System, das sperrige Helix -Verzerrungs -DNA -Läsionen erkennt und sie entfernt. Andererseits ersetzt die Reparatur von Nichtübereinstimmungen während der Replikation missbindete Basen. Der wichtigste Unterschied zwischen Reparaturenfehlerreparaturen und Reparatur von Nucleotid -Exzision besteht darin Die Nucleotid -Exzisionsreparatur (NER) wird verwendet, um Pyrimidin -Dimere zu entfernen, die durch UV -Bestrahlung und sperrige Helix -Läsionen durch chemische Addukte verursacht werden, während das Fehlpaarungsreparatursystem eine wichtige Rolle bei der Korrektur von Mehlbasis von Missbrauchs von Missverständnissen spielt, die aus Replikationsenzymen entkommen sind (DNA -Polymerase 1) während der Überporeplikation während der Postreplikation. Zusätzlich zu nicht übereinstimmenden Basen können MMR -Systemproteine auch die Insertions/ Deletions -Schleifen (IDL) reparieren, die Ergebnisse des Polymerase -Schlupfes während der Replikation von sich wiederholten DNA -Sequenzen sind.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist nicht übereinstimmende Reparaturen
3. Was ist die Reparatur von Nucleotid -Exzisionsreparaturen
4. Nebeneinander Vergleich - Reparatur von Nichtübereinstimmung gegen Nucleotid -Exzisionsreparatur
5. Zusammenfassung
Was ist die Reparatur von Nucleotid -Exzisionsreparaturen?
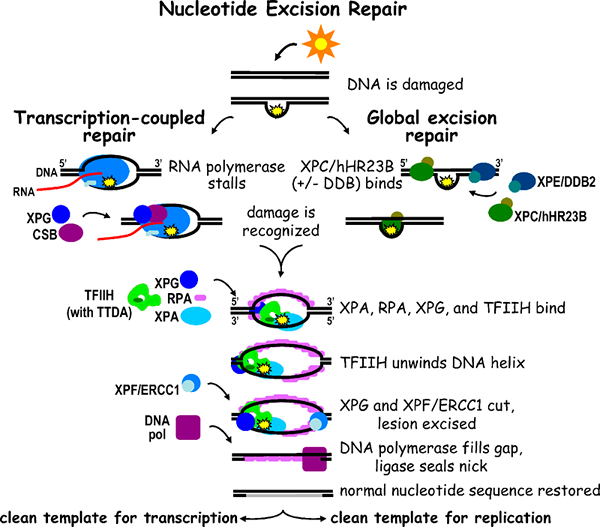
Das am stärksten angesehene Merkmal der Nucleotid -Exzisionsreparatur ist, dass es die modifizierten Nukleotidschäden repariert, die durch signifikante Verzerrungen in der DNA -Doppelhelix verursacht werden. Es wird in fast allen Organismen beobachtet, die bis heute untersucht wurden. UVR A, UVR B, UVR C (Exzinukleasen) UVR D (eine Helikase) sind die am bekanntesten am NER beteiligten Enzyme. UVR ABC Multi-Subunits-Enzymkomplex erzeugt die UVR A-, UVR B-, UVR-C-Polypeptide. Die für die oben genannten Polypeptide codierten Gene sind UVR A, UVR B, UVR C. UVR A- und B -Enzyme erkennen gemeinsam die von der DNA -Doppelhelix verursachte schadeninduzierte Verzerrung wie Pyrimidin -Dimmer aufgrund von UV -Bestrahlung. UVR A ist ein ATPase -Enzym und dies ist eine autokatalytische Reaktion. Dann verlässt UVR A die DNA, während der UVR -BC -Komplex (aktive Nuklease) die DNA auf beiden Seiten der durch ATP katalysierten Schädigung spalten. Ein weiteres Protein namens UVR D, das von UVRD -Gen codiert wird. Dies hinterlässt eine Lücke in der DNA -Helix. Nachdem beschädigtes Segment herausgeschnitten wurde, bleibt im DNA-Strang ein 12-13 Nucleotidspalt. Dies wird durch das DNA -Polymerase -Enzym I gefüllt und der Nick wird durch die DNA -Ligase versiegelt. ATP ist in drei Schritten dieser Reaktion erforderlich. Der NER-Mechanismus kann auch bei den Säugetieren-ähnlichen Menschen identifiziert werden. Beim Menschen ist der hautenbedingte als Xeroderma pigmentosum bezeichnete Hauterkrankung auf die DNA -Dimere zurückzuführen, die durch UV -Bestrahlung verursacht werden. Die Gene XPA, XPB, XPC, XPD, XPE, XPF und XPG erzeugen Proteine, um DNA -Schäden zu ersetzen. Die Proteine der Gene XPA, XPC, XPE, XPF und XPG haben die Nuklease -Aktivität. Andererseits zeigen die Proteine von XPB- und XPD -Genen die Helikaseaktivität, die analog E coli.

Abbildung 01: Reparatur der Nukleotid -Exzision
Was ist nicht übereinstimmende Reparaturen?
Das Fehlpaarungsreparatursystem wird während der DNA -Synthese initiiert. Selbst mit der funktionellen € Untereinheit ermöglicht die DNA -Polymerase III den Einbau eines falschen Nukleotids für die Synthese alle 108 Basispaare. Mismatch -Reparaturproteine erkennen dieses Nukleotid, verärgern Sie es und ersetzen Sie es durch das richtige Nukleotid, das für den endgültigen Grad der Genauigkeit verantwortlich ist. Die DNA -Methylierung ist für MMR -Proteine entscheid. Die Methylierung von Adenin (A) Nukleotid in einem GATC -Motiv eines neu synthetisierten Strangs ist etwas verzögert. Andererseits hat der übergeordnete Strang -Adenin -Nucleotid im GATC -Motiv bereits methyliert. MMR -Proteine erkennen den neu synthetisierten Strang durch diesen Unterschied zum Elternstrang und beginnen die Reparatur von Nichtübereinstimmung in einem neu synthetisierten Strang, bevor er methyliert wird. Die MMR -Proteine lenken ihre Reparaturaktivität, um das falsche Nukleotid zu vermitteln, bevor der neu replizierte DNA -Strang methyliert wird. Die Enzyme mut h, mut l und muts, kodiert von Genen mut h, mut l, muts diese Reaktionen in ecoli. Mut s Protein erkennt sieben von acht möglichen Mismpatch -Basenpaaren mit Ausnahme von C: C und bindet an der Stelle der Mismatch in der Duplex -DNA. Mit gebundenen ATPs verbinden sich Mut L und Mut s später dem Komplex. Der Komplex translokiert nur wenige tausend Basispaare weg, bis es ein hämethyliertes GATC -Motiv findet. Die ruhende Nukleaseaktivität von Mut H -Protein wird aktiviert, sobald es ein hämethyliertes GATC -Motiv gefunden hat. Es spaltet den nicht methylierten DNA -Strang, der einen 5' -Nick am G -Nukleotid des nichthylierten GATC -Motivs (neu synthetisierter DNA -Strang) hinterlässt, ein Nick und einen Nick -Nick -Strang hinterlässt. Dann wird der gleiche Strang auf der anderen Seite der Fehlanpassung von Mut H geklaut. In den restlichen Schritten werden die kollektiven Wirkungen von UVR d a Helicase-Protein, Mut U, SSB und Exonuklease das falsche Nukleotid in der einzelsträngigen DNA verärgern. Die Lücke, die in der Exzision gebildet wird. Ein ähnliches System kann bei Mäusen und Menschen identifiziert werden. Die Mutation von menschlichem HMLH1, HMSH1 und HMSH2 ist an erblicher Nichtpolyposis -Dickdarmkrebs beteiligt, der die Zellteilung von Dickdarmzellen derguliert.
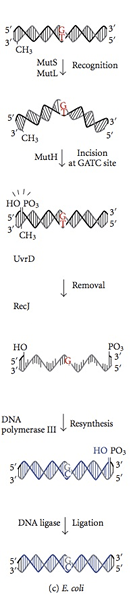
Abbildung 02: Nichtübereinstimmung Reparatur
Was ist der Unterschied zwischen der Reparatur von Nichtübereinstimmung und der Reparatur der Nukleotid -Exzision?
Reparaturfehlanpassungsreparatur gegenüber Nucleotid -Exzisionsreparaturen | |
| Das Fehlpaarungsreparatursystem erfolgt während der Nachbearbeitung. | Dies ist an der Entfernung von Pyrimidindimeren durch u beteiligt.V Bestrahlung und andere DNA -Läsionen aufgrund des chemischen Addukts. |
| Enzyme | |
| Es wird durch Mut S, Mut L, Mut H, Uvr D, SSB und Exonuklease i katalysiert. | Es wird durch UVR A, UVR B, UVR C, UVRD -Enzyme katalysiert. |
| Methylierung | |
| Es ist entscheidend, die Reaktion zu initiieren. | DNA -Methylierung ist nicht erforderlich, um die Reaktion zu initiieren. |
| Wirkung von Enzymen | |
| Mut H ist eine Endonuklease. | UVR B und UVR C sind Exonukleasen. |
| Gelegenheit | |
| Dies geschieht speziell während der Replikation. | Dies geschieht, wenn sie u ausgesetzt sind.V oder chemische Mutagene, nicht während der Replikation |
| Erhaltung | |
| Es ist hoch konserviert | Es ist nicht hoch konserviert. |
| Eine Lücke stopfen | |
| Dies geschieht durch DNA -Polymerase III. | Dies geschieht durch DNA -Polymerase I. |
Zusammenfassung - Reparaturenreparatur gegen Nucleotid -Exzisionsreparaturen
Mismatch Reparatur (MMR) und Nucleotid Exzisionsreparatur (NER) sind zwei Mechanismen, die in der Zelle stattfinden, um die DNA -Schäden und -verzerrungen zu korrigieren, die durch verschiedene Wirkstoffe verursacht werden. Diese werden gemeinsam als DNA -Reparaturmechanismen bezeichnet. Reparaturreparaturen der Nucleotid -Exzision Die modifizierten Nukleotidschäden, typischerweise die signifikanten Schäden der DNA -Doppelhelix, die aufgrund der Exposition gegenüber U auftreten.V Bestrahlung und chemische Addukte. Nichtübereinstimmungsreparaturproteine erkennen das falsche Nukleotid, verärgern es und ersetzen es durch korrektes Nucleotid. Dieser Prozess ist für den endgültigen Grad der Genauigkeit während der Replikation verantwortlich.
Referenz:
1.Cooper, Geoffrey M. „DNA -Reparatur.”Die Zelle: ein molekularer Ansatz. 2. Auflage.U.S. Nationalbibliothek für Medizin, 01. Januar. 1970. Netz. 09 März. 2017.
2.Mechanismen und Funktionen der DNA -Fehlpaarungsreparatur."Zellforschung. U.S. Nationalbibliothek für Medizin, n.D. Netz. 09 März. 2017.
Bild mit freundlicher Genehmigung:
1. „Nukleotid-Exzisionsreparatur-Journal.pbio.0040203.G001 ”von Jill O. Fuss, Priscilla K. Cooper - (CC von 2.5) über Commons Wikimedia
2. "DNA Mismatch Repair Ecoli" von Kenji Fukui - (CC von 4.0) über Commons Wikimedia