Unterschied zwischen Schrotflintensequenzierung und Sequenzierung der nächsten Generation
Der Schlüsselunterschied Zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation ist das Die Shotgun -Sequenzierung ist eine Sequenzierungsmethode, die DNA -Sequenzen zufällig in viele kleine Fragmente aufteilt und die Sequenz wieder zusammenstellt.
Die Sequenzierung ist der Prozess, der die genaue Reihenfolge von Nukleotiden in einem Gen, einer Gruppe von Genen, Chromosom und einem vollständigen Genom bestimmt. Es ist sehr wichtig in genomischen Studien, forensischen Studien, virologischen, biologischen systematischen, medizinischen Diagnose, Biotechnologie und in vielen anderen Bereichen, um die Struktur und Funktion von Genen und die Identifizierung von Organismen zu analysieren. Darüber hinaus sind verschiedene Arten von Sequenzierungsmethoden verfügbar. Shotgun -Sequenzierung und Sequenzierung der nächsten Generation sind zwei fortschrittliche Methoden unter ihnen.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist Schrotflintesequenzierung
3. Was ist Sequenzierung der nächsten Generation
4. Ähnlichkeiten zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation
5. Seite für Seitenvergleich - Schrotflintensequenzierung gegen Sequenzierung der nächsten Generation in tabellarischer Form
6. Zusammenfassung
Was ist Schrotflintesequenzierung?
Die Shotgun -Sequenzierung ist eine Sequenzierungsmethode, die die DNA. Im Allgemeinen sind Säugetiergenome strukturell komplex und größerer Größe. Daher sind sie durch Klonen schwer zu sequenzieren, da es zeitaufwändig ist. Die Schrotflintensequenzierung ist eine schnellere Methode. Es ist auch billiger, auszuführen. Daher verlassen sich moderne Wissenschaftler auf die Schrotflintensequenzierungsmethode, um komplexe Genome anzugehen.

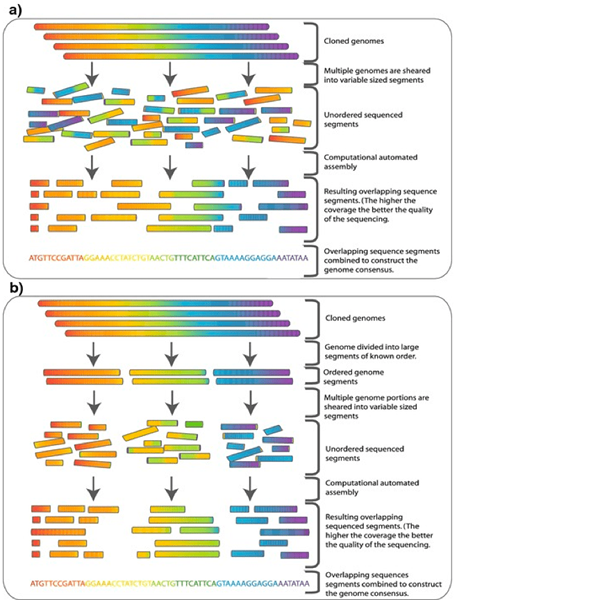
Abbildung 01: Schrotflintensequenzierung
Das Schrotflintensequenzierungsverfahren ist vergleichsweise einfach. Es beginnt mit der Fragmentierung des gesamten Genoms in unterschiedliche Größen von 20 Kilo-Basen bis zu 300 Kilo-Basen. Dann sollte jedes Fragment unter Verwendung der Kettenbeendungsmethode sequenziert werden. Nach der Sequenzierung ist es erforderlich, die Fragmente zusammenzustellen, indem Sie sich die überlappenden Regionen mithilfe einer ausgefeilten Computersoftware ansehen. Für diese Methode sind keine herkömmlichen Zuordnungen und Klonen von Sequenzen erforderlich. Darüber hinaus findet die Verwendung der genetischen Karte in dieser Methode nicht statt. Da jedoch keine vorhandenen Genomkarten verwendet werden, treten Fehler während der Anordnung wahrscheinlicher auf. Es ist eine der Hauptnachteile dieser Methode. Darüber hinaus liefern kürzere Fragmente weniger eindeutige Informationen für jede Lektüre in dieser Methode. Darüber hinaus erzeugt die Shotgun -Sequenzierung nicht genügend Daten, um eine Konsenssequenz nach dem erforderlichen Genauigkeitsstandard zu bestimmen.
Trotz der oben genannten Nachteile ist die Schrotflintensequenzierungsmethode derzeit die effizienteste und kostengünstigste Strategie zur Sequenzierung von mikrobiellen Genomen, einschließlich Bakterien, Viren und Hefe. Dies liegt daran.
Was ist Sequenzierung der nächsten Generation?
Die Sequenzierung der nächsten Generation (NGS) ist ein Begriff. Es beschreibt eine Reihe verschiedener moderner Sequenzierungstechnologien, die genomische Studien und molekulare Biologie revolutionierten. Diese Techniken umfassen Illumina -Sequenzierung, Roche 454 -Sequenzierung, Ionenprotonsequenzierung und feste Sequenzierung (Sequenzierung durch Oligo -Ligationserkennung). NGS -Systeme sind schneller und billiger. In NGS -Systemen werden vier HauptdNA -Sequenzierungsmethoden verwendet: Pyrosequenzierung, Sequenzierung durch Synthese, Sequenzierung durch Ligation und Ionen -Halbleitersequenzierung. Eine große Anzahl von DNA- oder RNA -Strängen (Millionen von) kann parallel durch NGs sequenziert werden. Es ermöglicht die Sequenzierung des gesamten Genoms von Organismen innerhalb eines kurzen Zeitraums.

Abbildung 02: Sequenzierung der nächsten Generation
NGS hat unterschiedliche Vorteile. Es ist ein hoher Geschwindigkeits-, genauerer und kostengünstigerer Prozess, der mit einer kleinen Stichprobengröße durchgeführt werden kann. Daher ermöglicht es die Analyse des gesamten menschlichen Genoms in einem einzelnen Sequenzierungsexperiment. Darüber hinaus können NGs in metagenomischen Studien, zum Nachweis von Variationen innerhalb eines einzelnen Genoms aufgrund von Insertionen und Deletionen usw. verwendet werden., und bei der Analyse von Genausdrücken. Darüber hinaus können NGs ganze Transkriptome aus einer großen Anzahl von Geweben gleichzeitig analysieren. Daher hat NGS die Analyse von Transkriptomen revolutioniert.
Was sind die Ähnlichkeiten zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation?
- Schrotflintensequenzierung und Sequenzierung der nächsten Generation sind zwei Genomsequenzierungsmethoden.
- Beide Methoden sind schnelle Methoden.
- Darüber hinaus sind sie kostengünstige Methoden.
- Sie können viele DNA -Fragmente parallel sequenzieren.
Was ist der Unterschied zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation?
Schrotflintensequenzierung und Sequenzierung der nächsten Generation sind zwei fortschrittliche Sequenzierungstechniken. Die Schrotflintensequenzierungsmethode unterteilt die DNA -Sequenzen des gesamten Chromosoms oder des gesamten Genoms zufällig in viele kleine Fragmente und stellt die Sequenzen durch, indem sie die überlappenden Sequenzen oder Regionen beobachten. Im Gegensatz dazu ist Sequenzierung der nächsten Generation (NGS) ein Begriff, der sich auf moderne Sequenzierungsprozesse mit hohem Durchsatz bezieht. Dies ist also der wichtigste Unterschied zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation. Darüber hinaus ist die Sequenzierung der nächsten Generation eine Technik, die von der Kapillarelektrophorese abhängt, während die Schrotflintensequenzierung nicht abhängt. Daher ist dies auch ein Unterschied zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation. Darüber hinaus ist die Sequenzierung der nächsten Generation im Vergleich zur Schrotflintensequenzierung hochempfindlich und sehr genau .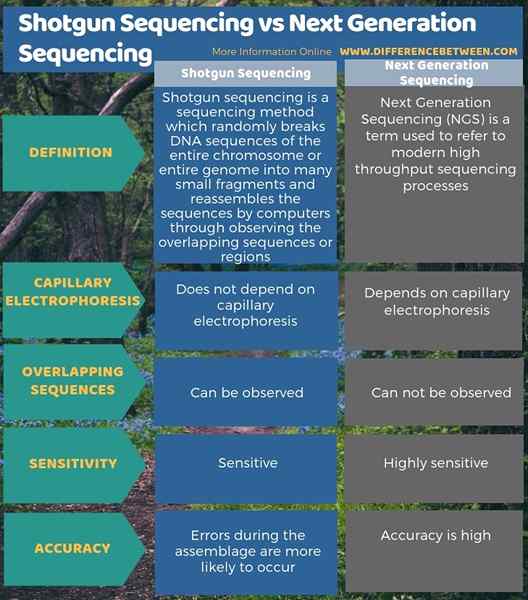
Zusammenfassung -Shotgun -Sequenzierung im Vergleich zur Sequenzierung der nächsten Generation
Sequenzierung und Sequenzierung der nächsten Generation sind zwei Sequenzierungsmethoden, die bei der Genomsequenzierung verwendet werden. Beide Methoden sind schnelle und kostengünstige Methoden. NGs wirkt nach einem Sequenzierungssystem nach dem Prinzip der Sequenzierung von Millionen von Sequenzen gleichzeitig schnell. Im Gegensatz dazu erfordert die Schrotflintensequenzierung das Aufbrechen von Genomen in kleine Fragmente und Sequenzierung und Wiederaufnahme unter Verwendung von überlappenden Sequenzen. Dies fasst den Unterschied zwischen der Schrotflintensequenzierung und der Sequenzierung der nächsten Generation zusammen.
Referenz:
1. "Komplexe Genome: Shotgun Sequencing" Nature News, Nature Publishing Group, hier verfügbar.
2. Behjati, Sam und Patricks Tarpey. „Was ist die Sequenzierung der nächsten Generation?Archive der Krankheit in der Kindheit. Ausgabe für Bildung und Praxis, BMJ Publishing Group, Dez. 2013, hier erhältlich.
Bild mit freundlicher Genehmigung:
1. "Ganzes Genom -Schrotflintensequenzierung versus hierarchische Schrotflintensequenzierung" von Commins, J., Toft, c., Tarife, m. A. - „Computerbiologische Methoden und ihre Anwendung auf die vergleichende Genomik endozellulärer symbiotischer Bakterien von Insekten.”Biol. Verfahren online (2009). Zugriff über Springerimages (CC BY-SA 2.5) über Commons Wikimedia
2. "Hiseq 2000" von Re73 - eigene Arbeit (CC BY -SA 3.0) über Commons Wikimedia