Was ist Unterschied zwischen nicht homologen Endverbindung und homologe direkte Wiederholung
Der Schlüsselunterschied zwischen nicht homologen Endverbindung und homologe direkte Wiederholung ist, dass das Nicht-homologe Endverbinden ein Weg ist, der doppelte Strangbrüche in DNA repariert, für die keine homologe Vorlage zur Reparatur erforderlich ist.
Die DNA -Reparatur ist ein Prozess, bei dem eine Zelle Schäden an den DNA -Molekülen identifiziert und korrigiert. Im Allgemeinen können normale Stoffwechselaktivitäten und Umweltfaktoren wie Strahlung DNA -Schäden verursachen. Diese Faktoren können zu Zehntausenden einzelner molekularer Läsionen pro Zelle und Tag führen. DNA-Doppelstrangbrüche Reparaturwege sind DNA-Reparaturwege in biologischen Zellen. Es gibt zwei DNA-Doppelstrangbrüche Reparaturwege als nicht-homologe Endverbindung und homologe direkte Wiederholung.
INHALT
1. Überblick und wichtiger Unterschied
2. Was ist nicht homologe Ende miteinander aus
3. Was ist homologe direkte Wiederholung
4. Ähnlichkeiten - Nicht -homologe Endverbindung und homologe direkte Wiederholung
5. Nicht -homologe Endverbindung gegen homologe direkte Wiederholung in tabellarischer Form
6. Zusammenfassung -Nicht -homologe Ende, die gegen homologe direkte Wiederholung zusammen ist
Was ist nicht homologe Ende miteinander aus?
Nicht -homologe Endverbindung (Nhej) ist ein Weg, der doppelte Strangbrüche in DNA repariert und keine homologe Vorlage benötigt, um die Reparatur zu leiten. Dieser Weg wurde 1966 von Moore und Haber gefunden. Dieser Weg wird typischerweise von kurzen homologen DNA-Sequenzen (Mikrohomologien) geleitet. Wenn die Überhänge kompatibel sind, repariert der NHEJ -Weg die Doppelstränge genau. Wenn die Überhänge jedoch nicht perfekt kompatibel sind, führt dies zu einer ungenauen Reparatur, die den Verlust von Nukleotiden verursacht. Der unangemessene NHEJ -Weg kann zu Translokationen, Telomerfusionen und Kennzeichen von Tumorzellen führen.
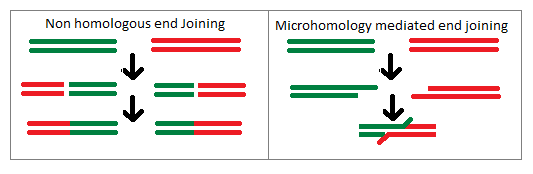
Abbildung 01: Nicht -homologe Endverbindung
Der NHEJ -Weg hat drei Hauptschritte: Endbindung und -bindel, Endverarbeitung und Ligation. Bei Säugetieren sind Proteine namens MRE11-Rad50-NBS1 (MRN), DNA-PKCs, KU (Ku70 und 80) an der Endbrücke beteiligt. Der Endverarbeitungsschritt beinhaltet die Entfernung von nicht übereinstimmenden oder beschädigten Nukleotiden und die Resynthese der DNA durch DNA-Polymerasen (Gap-Füllen). Die Entfernung von nicht übereinstimmenden oder beschädigten Nukleotiden wird durch Nukleasen wie Artemis durchgeführt. Die X -Familie -DNA -Polymerasen pol λ und μ in Säugetieren führen die Spaltfüllung durch. Die Endverarbeitung ist nicht erforderlich, wenn die Enden bereits kompatibel sind und 3'Hydorxyl- oder 5'phosphat -Termini haben. Darüber hinaus wird der endgültige Ligationsschritt mit dem Ligationskomplex IV durchgeführt, der aus DNA -Ligase IV und seinem Cofaktor XRCC4 besteht.
Was ist homologe direkte Wiederholung?
Homologe Direct Repeat (HDR) ist ein Weg, der doppelte Strangbrüche in DNA unter Verwendung einer homologen Vorlage zur Führung der Reparatur repariert. Der häufigste Weg der homologen direkten Wiederholung ist die homologe Rekombination. Der HDR -Mechanismus ist nur möglich, wenn im Kern ein homologe DNA -Stück vorhanden ist, hauptsächlich in der G2- und S -Phase des Zellzyklus. Der biologische Weg von HDR beginnt mit der Phosphorylierung von Histonprotein, der als H2AX in der Fläche bezeichnet wird, in der DNA-Doppelstrangbruch auftritt. Dies zieht andere Proteine an den beschädigten Ort an. Dann bindet der mRN -Komplex an beschädigte Enden und verhindert Chromosomenbrüche. Der mRN -Komplex hält auch zerbrochene Enden zusammen. Später werden DNA-Enden so verarbeitet, dass unnötige Residuen chemischer Gruppen entfernt und einzelne Überhänge gebildet werden.

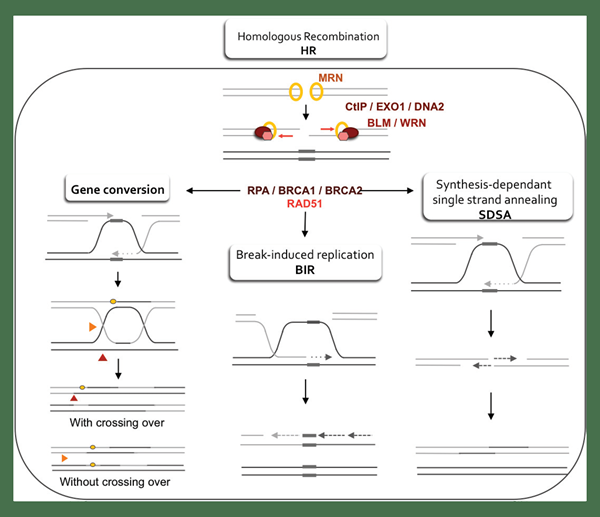
Abbildung 02: Homologe direkte Wiederholung
Jedes Stück einer einzelsträngigen DNA wird vom Protein bezeichnet, das RPA genannt wird, und seine Funktion besteht darin, einsträngige DNA-Teile stabil zu halten. Danach ersetzt Rad51 das RPA -Protein. Darüber hinaus passt RAD51 bei der Zusammenarbeit mit BRCA2 ein komplementäres DNA -Stück, das in den gebrochenen DNA -Strang eindringt. Die DNA -Polymerase wird durch ein anderes Protein, das als PCNA bekannt ist, an DNA gehalten. Letztendlich synthetisiert die Polymerase den fehlenden Teil des gebrochenen Strangs. Wenn der zerbrochene Strang neu synthetisiert wird, müssen beide Stränge wieder entkoppeln. Modelle für zahlreiche Möglichkeiten der Entkopplung werden vorgeschlagen. Nachdem Stränge getrennt sind, ist der Prozess abgeschlossen.
Was sind die Ähnlichkeiten zwischen nicht homologen Endverbiegungen und homologen direkter Wiederholung?
- Nicht-homologe Endverbindung und homologe direkte Wiederholung sind zwei DNA-Doppelstrangbrüche Reparaturwege.
- Der MRN -Komplex ist an beiden Wegen beteiligt.
- Nukleasen sind an beiden Wegen beteiligt.
- DNA -Polymerasen sind an beiden Wegen beteiligt.
- Diese Mechanismen finden sich sowohl in Prokaryoten als auch in Eukaryoten.
- Sie sind beide entscheidende Mechanismen für das Überleben von Zellen.
Was ist der Unterschied zwischen dem nicht homologen Endverbinden und der homologen direkten Wiederholung?
Das Nicht-homologe Endverbinden ist ein Weg, der doppelte Strangbrüche in DNA repariert, für die keine homologe Vorlage erforderlich ist, um die Reparatur zu lenken. Dies ist daher der wichtigste Unterschied zwischen nichthomologischem Endverbinden und homologen direkter Wiederholung. Darüber hinaus ist die homologe Rekombination nicht am nicht -homologen Ende der Beiträge beteiligt, während die homologe Rekombination an der homologen direkten Wiederholung beteiligt ist.
Die folgende Infografik stellt die Unterschiede zwischen nichthomologen Endverbindungen und homologer direkter Wiederholung in tabellarischer Form für Seite und Seitenvergleich dar.
Zusammenfassung -Nicht -homologe Ende, die gegen homologe direkte Wiederholung zusammen ist
Die DNA-Reparatur kann durch verschiedene Mechanismen wie direkte Umkehrung, Reparatur von Einzelstrangschäden, Reparaturen der Doppelstrang und die Translesionssynthese durchgeführt werden. Nicht-homologe Endverbindung und homologe direkte Wiederholungen sind zwei DNA-Doppelstrangbrüche Reparaturwege. Nicht homologe Endverbindung erfordert keine homologe Vorlage, um den DNA -Reparaturweg zu leiten. Homologe direkte Wiederholung ist ein Weg, der eine homologe Vorlage erfordert, um die DNA -Reparatur zu leiten. Dies ist also der wichtigste Unterschied zwischen nichthomologischem Endverbinden und homologen direkter Wiederholung.
Referenz:
1. „Nicht-Homologen-End-Zusammenarbeit.”Ein Überblick | Sciencedirect -Themen.
2. „Homology Regie Reparatur.”Ein Überblick | Sciencedirect -Themen.
Bild mit freundlicher Genehmigung:
1. "Nicht homologe End-Beitritt und mikrohomologisch vermittelte End-Joining" von CYNTH3004 bei English Wikipedia (CC BY-SA 3.0) über Commons Wikimedia
2. "Doppelstrang Break-Reparaturmodelle, die über homologe Rekombination wirken" von Camille Gelot, Indiana Magdalou und Bernard S. Lopez - https: // doi.org/10.3390/Genes6020267 (CC von 4.0) über Commons Wikimedia